Plataformas
Grupo de Biologia Computacional
e Bioinformática (GC2B)
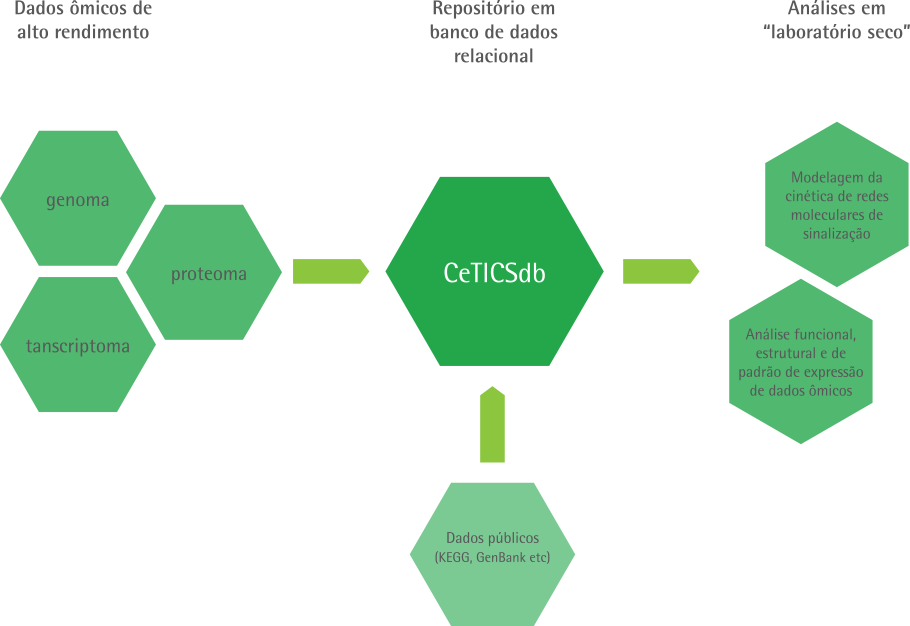
Os sub-projetos do CeTICS são largamente baseados na grande quantidade de dados biológicos que é produzida através de modernas técnicas de alto rendimento em ômicas (e.g., genômica, proteômica, transcriptômica). Dessa forma, visando a produção de conhecimento científico, existe uma demanda para organização de dados biológicos, assim como de sua recuperação, integração e análise. Bioinformática é o campo científico que lida com as demandas acima mencionadas, e inclui métodos para lidar com problemas que surgem nos sub-projetos do CeTICS, tais como: montagem, anotação e análise de genomas e de transcriptomas; identificação computacional de proteínas; análises taxonômica e filogenética; química computacional e modelagem molecular.
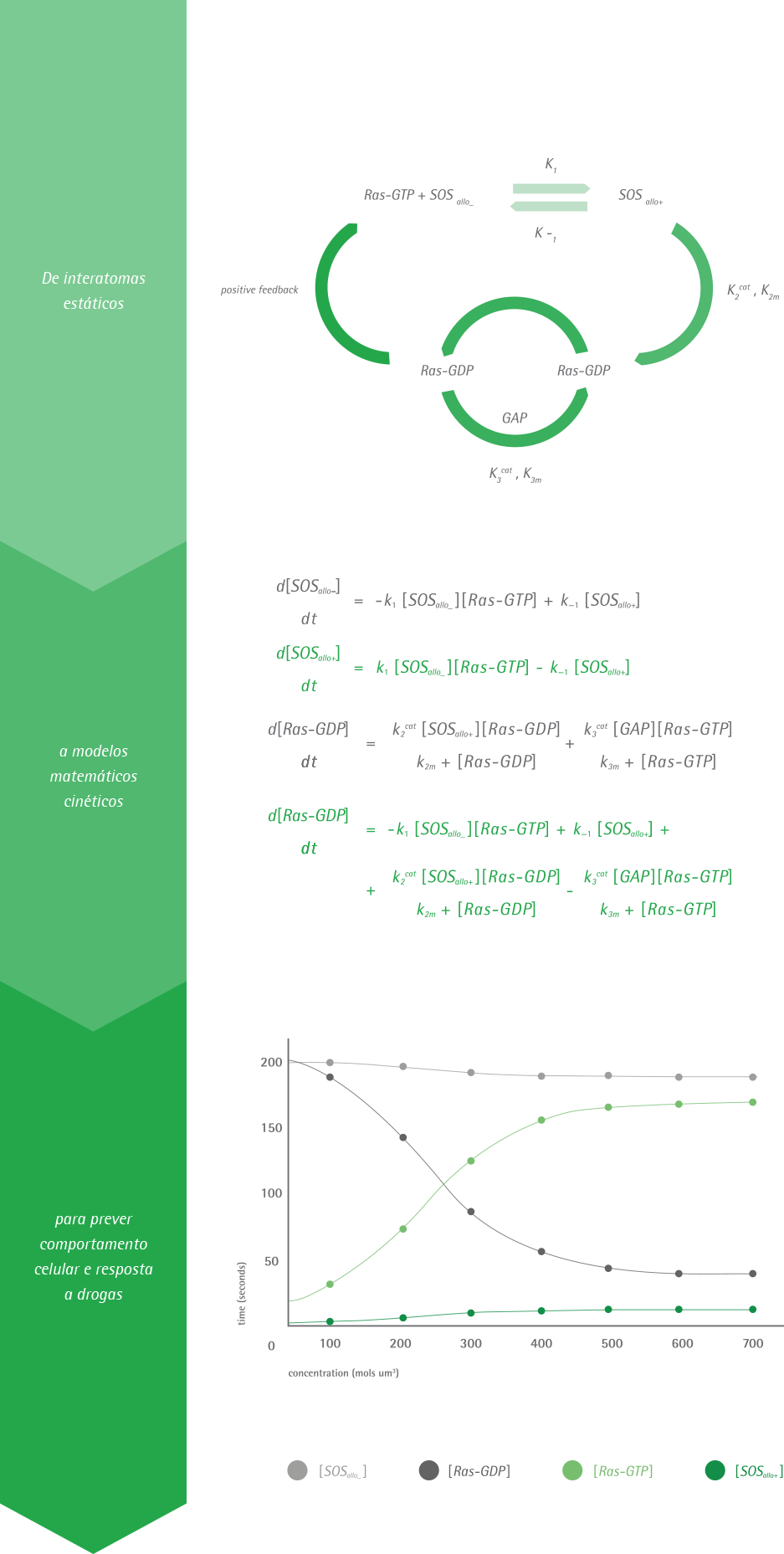
Por outro lado, os sub-projetos do CeTICS também estão baseados em investigações que são intrinsecamente interdisciplinares, uma vez que elas envolvem o cruzamento de fronteira entre disciplinas tais como Biologia Molecular, Bioquímica, Matemática, Estatística e Ciência da Computação. Essas investigações interdisciplinares, por sua vez visam o desenvolvimento de modelos matemáticos e a aplicação de técnicas de simulação computacional para estudar o comportamento de sistemas biológicos (e.g., a cinética de redes moleculares de sinalização), no contexto do campo científico conhecido como Biologia Computacional.
O Grupo de Biologia Computacional e Bioinformática (Group of Computational Biology and Bioinformatics – GC2B) foi criado no final de 2013, logo após o estabelecimento do próprio CeTICS. Ele tem como objetivo geral desenvolver e executar projetos científicos em Biologia Computacional e Bioinformática. Além disso, o GC2B tem um objetivo mais específico, que é prover a necessária assistência aos demais sub-projetos deste centro e também aos demais laboratórios do Instituto Butantan. Para este fim, alguns dos projetos que tualmente são executados pelo GC2B incluem o desenvolvimento da plataforma CeTICSdb para armazenamento e análise de dados ômicos heterogêneos [1,5], e o arcabouço SigNetSim para modelagem matemática e simulação computacional da cinética de redes moleculares de sinalização [2,3,4].
Equipe
- Cientistas do grupo
Hugo Aguirre Armelin
Junior Barrera
Kerly Fernanda Mesquita Pasqualoto
Marcelo da Silva Reis
Milton Yutaka Nishiyama Junior
Bioinformática
Recursos Computacionais
-

Servidora “CeTICS”
Dell PowerEdge R815 64
núcleos / 256 Gb RAM
24 Tb em 8 discos -

Servidora “Genomics”
Dell PowerEdge R815 32
núcleos / 129 Gb RAM
24 Tb em 24 discos